通过整合体外和计算机实验预测和阐明3D打印癌症细胞在水凝胶结
2024-05-28159区别3D生物打印与其他3D细胞培养技术的一个关键特征是其对创建结构的精确控制。这种特性 允许高分辨率制造具有可控结构和机械性能(如孔隙度、渗透性和刚度)的仿生结构。然而,分析打印 后的细胞动力学并优化其在3D制造环境中的功能只能通过反复试验和复制几个实验来实现。这个问题 促使了细胞自动机模型的shou次开发,以模拟3D生物打印结构中的打印后细胞行为。为了改进我们的模 型,我们使用MDA–MB–231细胞负载水凝胶生物打印了一个3D结构,并在11天内评估了细胞功能,包 括活力和增殖。结果表明,我们的模型成功地模拟了生物3D打印结构,并捕获了体外观察结果。我们 证明了该硅模型可以预测和阐明不同初始细胞数量的生物链接和不同的生物链接配方的明胶和海藻酸 盐打印后的生物学功能,而无需重复几个昂贵和耗时的体外测量。我们相信这样的计算框架将极大地 影响3D生物打印的未来应用。我们希望这项研究能够激励研究人员进一步认识到如何利用硅模型来推 进体外3D生物打印研究。
三维(3D)生物打印是新兴的3D生物制造方法之一,它被广泛应用于再生医学和组织工程中, 以制造复杂的模拟组织结构1。该技术在个性化治疗中具有巨大的应用潜力,更侧重于控制药物释放、癌症治疗药物筛选、研究可能的副作用以及分析肿瘤细胞的转移和侵袭2 。3D生物打印技术将细胞、生 物材料和受控运动系统结合起来,开发复杂的3D结构,并对结构的力学性能、孔隙度、渗透性和刚度 等特征进行精确控制3 –5。该技术通过结合细胞栖息地的重要方面,可以克服传统3D方法的多个限制。这些方面包括类似于肿瘤的天然细胞外基质(ECM)的非均匀3D微环境,细胞与邻近细胞和局部ECM 的 复杂相互作用,以及营养物质和氧气的复杂扩散过程6– 8。因此,这种方法可以更好地代表细胞生长机 制的见解,并提供体内肿瘤动力学和癌细胞对治疗反应的更紧密预测7。尽管3D生物打印技术发展迅速,但仍有一些挑战需要解决。目前,生物打印技术主要是在试错 的基础上实现预期的输出,增加了对实验技术的需求。这种反复试验的基础包括优化生物墨水的性能 及其印刷性、结构机械强度和打印期间和打印后的细胞活力9。因此,优化生物打印相关的实验是非常 昂贵的。这些挑战使得实验设计和数据收集在这个过程中变得更加复杂。
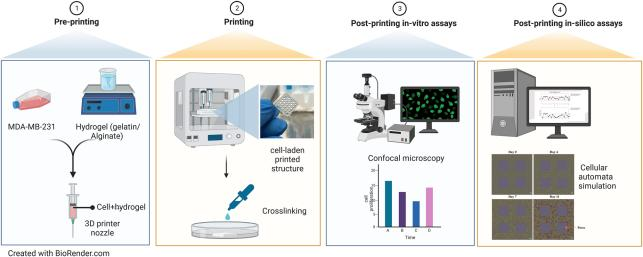
图1所示。本研究涉及的主要步骤示意图;步骤1:制备由明胶、海藻酸盐和MDA-MB-231细胞系组成 的生物链;步骤2:打印和交联承载细胞的3D结构;步骤3:进行印后体外检测;步骤4:开发和校准 一个硅模型。
硅内方法可用于补充体外实验并协助解决该3D方法的一些局限性10–12。常见的内部方法包括机器 学习(ML)方法和机制建模。机器学习模型可以进一步分类为深度神经网络、随机森林、支持向量机(SVM)和树分类器。机器学习越来越多地应用于3D打印过程的不同阶段,如工艺优化、施工精度分析 、缺陷诊断和生物墨水性能预测13。例如,Xu等人14成功地使用ML方法创建了一个预测模型,以良好 的灵敏度预测细胞活力,并评估各种工艺参数(包括紫外线强度和紫外线曝光时间)对基于立体光刻的 3D生物打印中细胞活力的重要性。多项研究也试图开发不同的基于ml的技术来优化生物墨水的可打印 性。例如,Lee等人15使用多元回归分析证明了印刷适性与油墨机械特性之间的关系。然而,尽管ML模 型在生物医学领域的应用取得了很大的进展,但由于数据收集的过程和研究的目的,这种方法面临着 一些局限性。
为了做出准确的预测,机器学习模型需要大量的数据,选择合适的算法,并确定感兴趣的输入和输出16。从生物打印研究中获取大量数据可能是不切实际的,这些研究涉及昂贵的细胞和材料以及 耗时的过程。ML应用程序的另一个常见挑战是它们可能难以解释。这可能会导致寻求生物打印结构中 细胞行为机制解释的研究人员缺乏信任17。相比之下,机制建模关注的是通过对潜在机制的假设来描 述现象。机制模型包括随机和基于主体的模型、离散模型、常微分方程(ode)和偏微分方程(PDEs)。这 种机制模型可以提供洞察生物打印结构中细胞行为的机制方面。因此,它们是我们在这项研究中shou选 的数学技术。
作为生物3D打印过程的一部分,对机械建模和仿真的研究越来越多。例如,有多项数学研究预 测了施加在喷嘴中的生物链接和细胞上的剪切应力18,19 ;基于生物墨水材料性能的z终打印结构力学性 能预测20;并模拟生物链接的沉积过程和3D结构的z终形状9。这些数学技术试图模拟生物打印过程-在 生物打印过程中和之后-研究不同参数对生物打印的各个方面的影响,包括细胞活力和结构稳定性。
然而,关于细胞嵌入生物3D打印结构的打印后行为的全面计算研究尚未报道。这样的数学研究 可以为研究人员提供更多关于打印后细胞功能的见解,并允许他们预测复杂的细胞活动和优化细胞微 环境,通过将实验评估保持在z低限度来节省金钱和时间。这是第一项旨在开发体外和硅生物3D打印 乳腺癌模型的整合研究,以研究嵌入生物3D打印结构中的乳腺癌细胞群的打印后行为(图1)。在体外研究中,我们使用了MDA-MB-231细胞系,这是癌症相关研究中z常用的z具侵袭性的乳腺癌细胞 系之一。为了开发生物墨水,选择明胶和海藻酸盐的混合物作为3D生物打印中z常见的生物墨水材料,因为它具有与天然ECM相似的特性,并且具有适合生物打印应用的生理和生物学特性21。在我们的计算机研究中,我们选择了元胞自动机建模,其中大部分参数都是从实验数据中选择的。为了提高模 型的可靠性,还进行了全面的体外实验。
本研究中提出的基于主体的模型有利于展示复杂的细胞系统,包括细胞增殖、运动、细胞与环 境的相互作用(例如,ECM、邻近细胞和资源消耗)以及支架内的细胞聚集。在这项研究中,我们证 明了数学模型和计算机模拟可以用来捕捉体外动力学。这种体外和芯片研究的结合可以提高研究人员 对3D生物打印结构中细胞活动的理解,从而加速和提高优化和实验设置的准确性。所提出的硅模型是 非常有前途的,并能够进一步发展,应用于3D细胞培养使用生物打印技术在不同的生物医学应用。
结果与讨论
体外细胞研究。成功打印出肿瘤样水凝胶网络模型(图2a)。为了确定包埋在水凝胶中的MDA-MB- 231细胞的增殖情况,采用MTT法监测细胞在第0、4、7、10和11天的代谢活动。如图2b所示,与第0天 相比,3D细胞/水凝胶构建的MDA-MB-231细胞在第4、7、10和11天分别表现出1.86倍、2.7倍、2.78倍 和2.8倍的增殖。确实,细胞在前7天表现出快速增殖,从第7天到第11天几乎保持了几乎稳定的细胞 增殖。有趣的是,结果表明,在3D微环境中封装的MDA-MB-231的倍增时间比在2D培养中生长的细胞高 3倍,这可以归因于3D基质中细胞活性的降低22。从第7天到第11天,增殖细胞的数量大致保持不变,留下了一个问题:细胞是死亡还是由于不希望的条件进入非增殖期(静止期)。为了回答这个问题, 在11天的时间里,我们进一步采用了活死实验和Ki-67免疫染色,以更好地了解包裹在3D支架中的细 胞行为生长采用活死染色法观察MDA-MB-231 在11天内的生存能力,结果与 MTT试验结果。如图2所示,打印 后大部分细胞都能存活,第0天细胞存活率为76±2%,这清楚地表明生物打印过程对细胞活力的损害 较小。第1周存活率逐渐提高,第4天和第7天分别达到98±1%和99±1%。因此,该结构具有足够的多 孔性,使氧气和葡萄糖能够通过水凝胶支架扩散和分布,为细胞提供适宜的生存环境。从第7天到第1 1天,虽然有部分细胞死亡,但大部分细胞存活,第11天存活率达到96±2%。通过对比活死实验和MTT 实验的结果,可以得出结论,在支架达到z大容量的7天后,有相当一部分细胞进入了静息期,部分 细胞在长期静息期开始死亡,或者是由于缺乏资源。本实验中的细胞死亡几乎可以忽略不计,这说明 明胶/海藻酸盐支架在生物打印中的应用前景广阔。
为了观察MDA-MB-231的增殖能力,将细胞固定,并使用抗Ki-67抗体在共聚焦显微镜下对增殖细 胞进行成像(图3,4)。Ki-67是一种常用的标记物,在细胞周期的所有活跃阶段都存在,但在细胞 的固定阶段则不存在23。结果表明,在第0天和第4天,ki-67的阳性细胞分别接近98±1%和95±2%,而 在第7天,这一数字下降到86±2%,然后在第11天急剧下降到约48.2±2.4。这一结果与前面的数据一 致(图3),说明在7天内,细胞不仅存活,而且保持了增殖能力。从第7天到第11天,尽管有很大比 例的细胞存活,但由于缺乏足够的细胞增殖空间,它们处于静止状态,无法再增殖。此外,在第7天 和第11天,细胞变得更加聚集,特别是靠近毛孔,细胞聚集中心的细胞由于被其他细胞包围而显示出 不增殖。因此,由于没有足够的空间放置子细胞,并且由于聚集体中心缺乏营养和氧气,增殖过程被 中止。
将体外实验结果用于参数化所建立的数学模型。
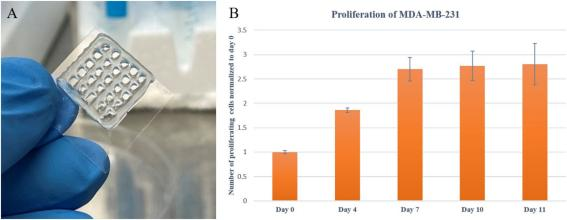
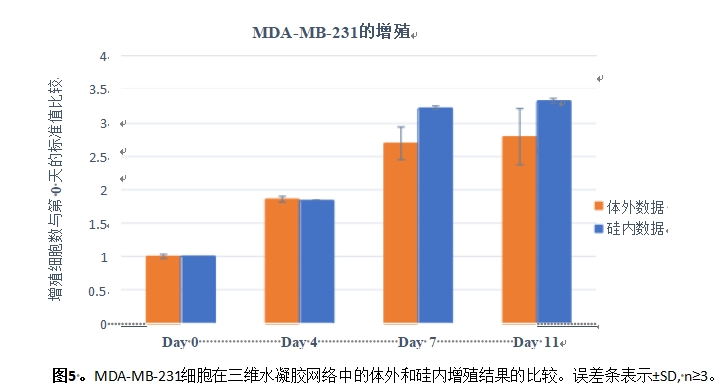
图2 。(A):利用生物3D打印技术制备细胞/水凝胶结构;(B):从第0天到第11天,MDA-MB-231在水 凝胶网络中包埋的增殖。误差条表示±SD, n≥3。
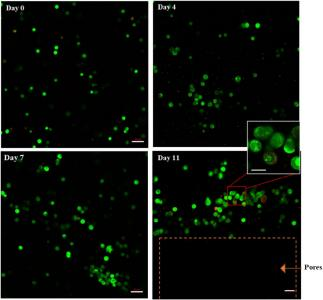
图3 。从第0天到第11天,使用荧光活/死检测试剂盒,显微镜图像显示了3D水凝胶构建中的MDA-MB- 231细胞的活力。分别用calcein-AM 和 PI 对活细胞和死细胞进行染色(绿色为活细胞;红色代表死细 胞)。使用激光扫描共聚焦显微镜对细胞进行成像。比例尺,50 μm(放大图像,比例尺,30 μm)。
硅细胞研究。使3D生物打印技术优于其他3D细胞培养技术的一个基本特性是它提供了对创建结构的精确控制。这种特性为制造具有可控结构和机械性能(如孔隙度、渗透性和刚度)的仿生结构提 供了机会24-26。然而,分析打印后支架内的细胞行为仅取决于执行不同的体外测量。实验测量生物3D打 印结构中细胞行为的某些方面是具有挑战性的,因为缺乏精确的定量技术,例如定义细胞-细胞和细 胞-微环境相互作用。这个问题促使我们开发了一个数学框架来模拟打印后支架内的细胞行为。这个 框架不仅要克服这些挑战,而且要在不重复实验的情况下准确地设计和预测打印后的细胞功能。
使用元胞自动机的基于个体的建模是在细胞水平上模拟细胞生长的时空机制的一种方法27,28。这 种方法是一个动态系统,包括细胞网格,每个细胞都有一组离散状态29。近年来,CA模型被广泛用于 研究基于各种静态自动机规则的不同癌细胞机制30。然而,到目前为止,还没有研究将CA建模应用于 使用3D生物打印技术3D培养癌细胞。我们选择这种数学方法来模拟封装在3D生物打印结构中的细胞生 长,因为它能够捕捉3D打印结构的空间特性,并且能够灵活地探索不同的假设。此外,由于从体外实 验中获得的数据包含离散形式的细胞,因此这种离散数学技术将更准确地模拟这一过程。本研究中开 发的框架代表了细胞增殖、活力、运动和与环境相互作用的规则,包括水凝胶和邻近细胞,以及水凝 胶网络内的簇形成。本文中演示的计算机结果基于n=100次模拟运行的平均值和标准偏差,其中n是由 一致性分析驱动的(补充材料,一致性分析)。
图5显示了在体外和在硅材料中支架内11天的细胞增殖模式,说明它们彼此一致。在细胞增殖 过程中,初始细胞密度和支架容量是我们分别指定为初始变量和C变量的两个关键参数。这些参数的 相应值通过校准来确定,以产生与体外研究的z佳拟合。请注意,所开发模型中的初始细胞密度代表 了薄层内可以相互作用的细胞的有效初始种群,而不是支架中细胞的总数。因此,与实验设置中的细 胞密度相比,模拟细胞密度减少了一个比例因子。结果表明,模拟细胞和实验细胞均在7天后达到z大细胞密度。细胞在支架中达到z大密度所需的时间在很大程度上取决于初始细胞的数量和打印支架 的容量。初始细胞数量越多,支架容量越小,细胞越早达到z大细胞密度,越早停止增殖。因此,通 过微调这些参数并在不同情况下进行模拟,研究人员可以设计实验以获得所需的结果,而无需重复体 外分析。
模拟数据也能够一致地复制活力和增殖实验结果。如前一节所述,虽然细胞在打印后7天内的存 活率约为99%,但在第7天至第11天,死亡细胞的数量略有增加,此时大部分细胞处于静止期。因此,
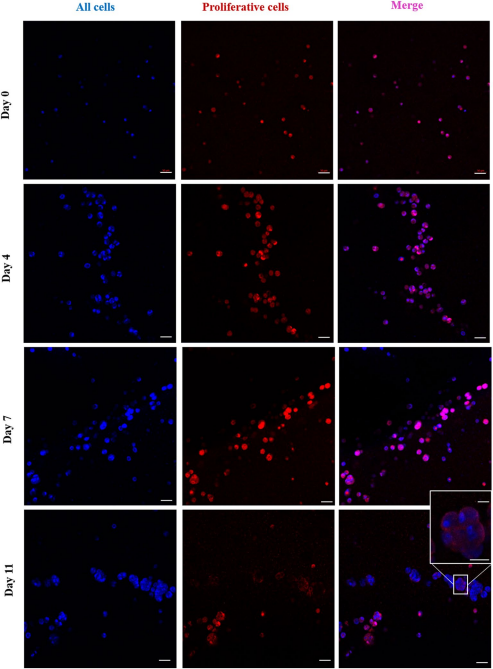
图4 。生物3D打印构建体中包裹的MDA-MB-231细胞的Ki-67染色。用Alexa Fluor 546和Hoechst 33 , 342标记的抗ki-67抗体对细胞进行染色(红色表示ki-67阳性细胞;蓝色表示所有单元格)。使用激光 扫描共聚焦显微镜对细胞进行成像。比例尺,50 μm(放大图像,比例尺,30 μm)
为了精确地模拟体外条件,我们假设细胞保持在一个延长的固定期超过指定的小时,由一个随机数字 (Cd)定义,开始以指定的概率(Pd)死亡。这种观察结果可以从生物学上解释为细胞在进入细胞静 止期后无法重新进入细胞周期。
图5显示了在水凝胶网络中生长的硅MDA-MB-231细胞的快照;以及细胞三维结构的体外显微图 像。在这张图中,你可以看到细胞分布和细胞团形成的进展在0 ,4 ,7和11天的体外和在硅。在计算机 图像中,黄色、红色和黑色细胞分别代表增殖、非增殖和死亡细胞。
第0天,少量细胞分布在水凝胶网络内。随着时间的推移,细胞增殖,产生了z初的两个细胞群 , 然后是更大的细胞群。与体外观察相似,到第11天,存活率保持在100%左右,第11天存活率略有下 降,为93.74±0.5%0。此外,模拟细胞增殖随着时间的推移而下降,并且在7天后由于达到支架的z大 容量而显著下降;z终在第11天降至54.14%±0.25%。11天水凝胶支架内细胞生长的动画也可在补充材 料中获得(图S3)。
除了细胞活力和增殖外,另一个重要因素是细胞在周围基质中移动的能力。这种模拟也可以应 用于分析细胞运动以及水凝胶网络中形成的肿瘤簇的结构和分布,而无需实验评估。肿瘤簇可能是由 于邻近细胞之间或亲本和子细胞之间的相互作用而产生的,这取决于它们的位置和微环境31。事实上, 细胞通过细胞间的物理和信号相互作用进行协调,并形成集群。
在图S1(补充材料)中,细胞表现出向支架孔爬行的趋势,然后在这些孔周围形成簇,因为必需 资源在那里更集中,特别是在7天后。这一事实表明,水凝胶网络具有有限的资源运输能力。因此,我 们定义了细胞-细胞信号(LC)和细胞-孔吸引(Lp)的特定吸引力范围,以考虑不同的细胞迁移方向。
运动速度是影响星团形成的另一个重要参数。Fallica等研究表明,由于材料刚性和细胞受体的锚定作 用,癌细胞在三维微环境中运动受到抑制,且运动速度极低22。因此,基于本研究和以往研究的观察, 我们对细胞运动速度进行了校准。在运动过程中,每个个体在特定时间(定义为mC)沿着基于细胞吸引 力确定的方向运动。在校准过程中,通过比较体外和计算机上的结果,得出的结论是,与支架表面/孔 隙(Lp)相比,在较小的吸引力范围(LC)内,细胞向邻近细胞移动的倾向较小。在模型中应用这些规则 (图6),细胞在运动和簇形成方面模仿体外细胞行为。该模型的结果与先前的研究一致22,32。
总的来说,该模型是为了结合体外3D生物打印评估而开发的,从而对整个3D制造结构进行全面 分析。该模拟的主要应用之一是预测在未实践的微环境中打印后的细胞行为,从而提高其复制所需生 物设置的能力。例如,该模型提供了评估不同重要参数(如各种初始细胞密度)在长期内对细胞行为的 影响的机会。这有利于研究人员在不需要重复实验的情况下产生更适合细胞生长的微环境。例如,他 们可以从大小或结构形状方面设计支架, 目的是修改支架的容量,以提高细胞增殖和减少细胞死亡。
硅学模型验证。为了进一步验证硅模型,我们在两种不同的情况下使用不同的实验变量进行生物打印过程:情况1:不同的初始细胞密度;案例2:不同的生物油墨配方。在案例1中,我们使用含有4% (w/ v)明胶、4% (w/v)海藻酸盐和1.5×1 06 MDA-MB-231细胞mL – 1的生物墨水进行生物打印。案例2:我 们使用终浓度为4% (w/v)明胶、5% (w/v)海藻酸盐和2×1 06 MDA-MB-231细胞mL – 1的生物墨水进行 生物打印实验。使用校准的计算机模型,我们想预测细胞在这两种新条件下的增殖模式。
图7显示了病例1在体外和在计算机上10天的细胞增殖模式,说明它们彼此一致。在硅模型中, Cinitial(初始细胞密度)外,所有参数与校准模型中的值相同。与实验设置中的细胞密度相比,模拟

图6 。中间的面板显示了MDA-MB-321在硅三维水凝胶结构中的生长情况;黄色代表增殖细胞;红色代表 非增殖细胞;黑色代表死细胞。左右两张图分别为第0天、第4天、第7天和第11天在相差显微镜下观察 到的包裹在3D水凝胶结构中的MDA-MB-231细胞:比尺,50 μm。
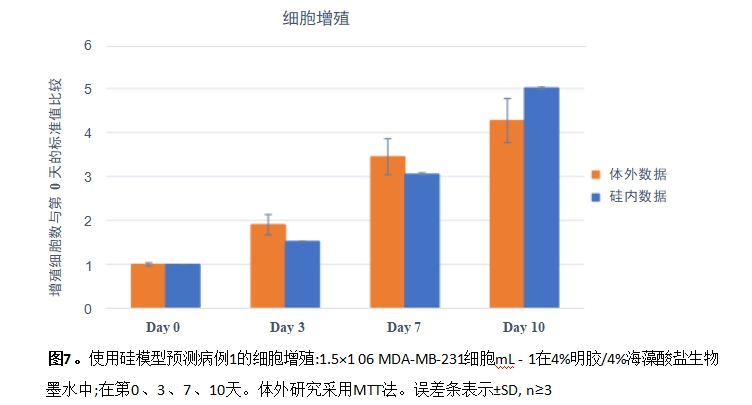
细胞密度减少了相同的比例因子,并设置为 Cinitial = 2000。模拟和实验均表明,7天后细胞密度 未达到z大,仍在继续生长。因此,当初始细胞数量减少时,后期细胞达到其支架能力并因此停止增 殖。
图8比较了病例2的体外和硅细胞增殖模式,也显示了一致性。在这个实验中,我们改变了生物 链接的配方。增加海藻酸盐浓度可以增加水凝胶结构的刚性,如前所述 33。还发现微环境的刚度也
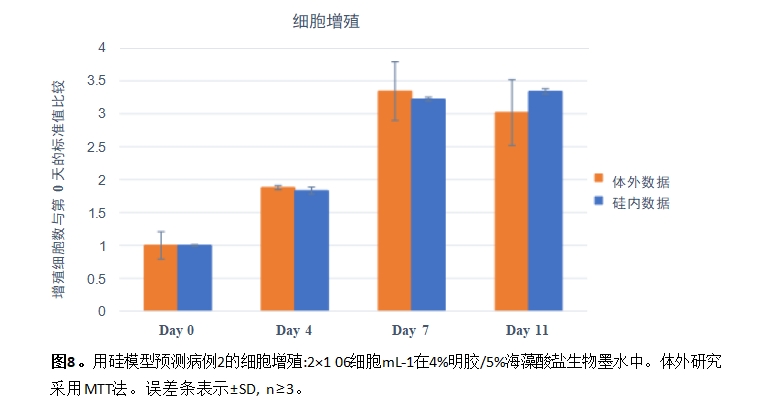
会影响支架内细胞的运动和球体的形成34。虽然与刚度直接相关的参数尚未整合到我们的模型中 , 但我们可以通过改变细胞运动的一些规则来调节细胞行为并研究生物链接配方和刚度对增殖和迁移 的影响。在含有4% (w/v)明胶和4% (w/v)海藻酸盐的生物墨水校准模型中,细胞每15小时移动一次, 用mc表示。因此,当含有4% (w/v)明胶和5% (w/v)海藻酸盐的生物墨水时,我们降低了细胞在更硬的 微环境中的移动速度,将mc改为20小时,同时保持其他参数不变。将计算机模型的结果与体外数据进 行比较,我们得出结论,对于4% (w/v)明胶和5% (w/v)海藻酸盐基生物链,mc=20 h与体外11天内的 增殖趋势非常接近。
体外观察显示,第11天,细胞增殖略有下降,这可能与微环境的刚性有关。事实上,这种刚性 可能会减少细胞的运动和增殖;并阻碍营养物质的运输,导致细胞随着时间的推移死亡。对于生物墨 水配方更显著的变化,有必要将微环境刚度或生物墨水相关参数纳入模型,以准确预测细胞行为。然 而,加入4% (w/v)明胶,5% (w/v)海藻酸盐,并对生物墨水配方进行少量修改,我们开发的模型可以 成功使用。
综上所述,我们可以通过在体外数据中创建变化来验证我们的模型,并成功地模拟了不同的情 况。因此,我们可以自信地说,这个模型可以帮助研究人员通过预测结果更准确地计划实验。事实上 , 研究人员在各种情况下进行模拟并微调相关参数,可以设计实验以达到所需的结果,而无需重复体外程序。
前景。本研究开发的数学框架可以通过扩展其规则和提高其提供生物系统准确预测的能力,广 泛应用于组织工程、肿瘤学和制药行业等不同应用的不同生物打印相关研究。我们的模型可以通过纳 入生物墨水相关参数(如刚度和结构完整性)来扩展,这些参数调节细胞行为,包括增殖和迁移以及氧 气/营养物质向3D网络的扩散35-39。
此外,所提出的模型可以与机器学习算法集成,并为研究人员提供了预测水凝胶网络对生物系 统中任何期望目标的时间或结构效应的机会。此外,我们可以使用CA模拟对ML算法进行预训练,然后 使用迁移学习方法对实验数据进行训练。
该模型的另一个应用前景是在多细胞系的异质环境中研究细胞-细胞相互作用和细胞- ecm相互 作用。此外,通过调整规则,该模型可以与药代动力学建模技术相结合,利用生物3D打印技术模拟3D 细胞培养过程中的药物治疗反应,以帮助研究肿瘤的发展和转移、药物筛选等癌症研究。z后,我们相信这项工作或其与其他建模技术的结合可以显著影响未来3D生物打印的发展,并 在很大程度上避免进行昂贵和耗时的实验
结论
到目前为止,在3D生物打印水凝胶中生长的细胞的z佳打印后行为仅通过复制几个实验实现,这些实 验既耗时又昂贵。为了克服这一挑战,我们开发了一个CA模型来模拟3D生物打印结构中的打印后细胞 动力学。为此,我们首先成功地打印了MDA-MB-231/明胶/海藻酸盐生物链接,并在11天内使用MTT,
Live-dead和Ki-67细胞增殖试验评估了细胞行为。利用体外实验结果,我们在CA模型中定义了3D水凝 胶网络中细胞增殖、活力、运动和簇形成的规则,并校准了模型参数,如加倍时间、运动速度和11天 内死亡概率。我们的模型可以定量捕捉3D支架中细胞在体外打印后的行为,并能够预测和阐明不同生 物打印条件下的细胞行为。例如,它复制了细胞运动和细胞向毛孔爬行的过程,然后在7天后由于营 养和氧气分布不均匀而形成集群。此外,计算机数据阐明了细胞增殖依赖于细胞的初始数量和打印的 水凝胶网络的容量,并可以根据生物墨水中细胞的初始数量预测打印后的细胞增殖。这种硅模型也可 以用含有明胶和海藻酸盐的各种网络配方来表示细胞活性。提出的数学框架可以帮助研究人员产生更 适合细胞生长的微环境,而无需重复实验。因此,我们的数学框架可以被认为是涉及生物打印相关研 究进步的一个足智多虑的步骤。
材料与方法
材料。从加拿大Sigma-Aldrich公司获得了褐藻酸钠盐、二甲亚砜 (DMSO)、氯化钠、氯化钙(CaCl2) 和牛皮明胶(B型)。对于细胞培养研究,MDA-MB-231购自ATCC, DMEM (Dulbecco ‘s Modified Eagle Medium)、FBS(胎牛血清)、青霉素/链霉素、胰蛋白酶/EDTA溶液0.25% (w /v)和磷酸缓冲盐水(PBS) 片购自Wisent Bioproducts 。 (3-(4,5-二甲基-2-噻唑)-2,5-二苯基- 2h -溴化四氮唑)MTT粉末、Triton X-100、牛血清白蛋白和多聚甲醛购自加拿大Sigma-Aldrich公司。此外,活/死细胞活力测定 试剂盒(CBA415), Hoechst 33342nucleus Dye 由Sigma-Aldrich(加拿大)提供。此外,抗ki67抗体(ab15580)和Alexa Fluor 546 山羊抗兔IgG (H+L)分别购自Abcam和Invitrogen(加拿大)。
细胞培养。MDA-MB-231细胞在T-75瓶中加入10%胎牛血清和1% 100 Uml-1青霉素/链霉素的 DMEM中培养。细胞在5% CO2和37℃条件下孵育,每隔一天更换一次培养基。当细胞达到 80%的合流度时,用 DPBS冲洗细胞两次,然后用胰蛋白酶/EDTA (0.25%-1X)悬浮。
生物打印。为了制备生物墨水,根据欧阳等人38提供的方案,首先用去离子水制备0.5% NaCl溶液。然后加入明胶和褐藻酸钠盐粉,剧烈搅拌 1 h。将配制好的溶液在70℃(30 min, 3次)加热灭菌, 4℃保存后使用。在生物打印之前,使用混合注射器将 MDA-MB-231悬浮液与制备好的明胶/海藻酸盐溶 液轻轻均匀混合,制成z终浓度为4% (w/v) 明胶,4% (w/v)海藻酸盐和2-2.5×1 06 MDA-MB-231 细胞 mL-1的生物墨水。
使用CELLINK INCREDIBLE+ 挤压生物打印机进行生物打印。将制备好的生物墨水用针挤压制成三 维细胞-水凝胶层状网格结构。具体来说,本实验使用的打印喷嘴为标准锥形喷嘴 (22G),针的移动速 度调节为5mm-1。印刷是在室温下施加适当的压力完成的。采用 Solidwork软件进行结构设计,在sli3r软件中切割成10层,尺寸为10 × 10 × 3mm3 ,采用直线填充模式。在打印过程中,我们努力 将压力保持在 z低水平,以使对细胞活力的损害z小。随后,将携带细胞的构建体浸泡在 3% (w/v) 无菌氯化钙溶液中(20分钟),使海藻酸钠与钙离子交联21。然后,PBS洗涤三次后,每个结构在含有 10%胎牛血清和1%青霉素/链霉素的DMEM培养基中培养于12孔板中,在5% CO2和37℃下孵育一段预定时 间。每隔一天更换一次培养基。
MTT试验。采用MTT法检测三维网络内细胞增殖。预定孵育天数后,去除含有 3D结构的12孔板中每孔中的培养基,在每孔中加入900µL新鲜培养基和100µL MTT溶液(0.5 mgmL – 1 in PBS ),在37℃CO2培养箱中孵育4 h。控制组是一个没有任何细胞的3D结构。然后,抽吸培养基后,在每孔中加入 1mlDMSO,在37℃的CO2培养箱中溶解形成的甲醛晶体30min。z后,利用Absorbance Microplate Reader 在540 nm处记录溶解后的甲醛晶体的强度。
活死检测。根据制造商的说明,使用活/死染色活力试剂盒在特定时间点对生物 3D打印的细胞负载构建体进行染色,以确定细胞活力。简单地说,每个构建体在 PBS中洗涤三次。然后,将1µM Calcein- AM和2µM碘化丙啶加入染色细胞,并在黑暗中孵育。使用激光扫描共聚焦显微镜 (蔡司LSM 700)对三维 结构内的活细胞和死细胞进行多点成像。然后使用 ImageJ软件对图像进行分析。细胞活力通过将绿色 (活)细胞的数量除以每张图像中的细胞总数来计算。
细胞增殖。Ki-67细胞增殖试验是一种评价体外细胞增殖的定量技术。使用这种方法,Ki-67蛋白被用 作细胞增殖的标记物,因为它在活跃的细胞周期(G1, S, G2和M)表达,而不是在固定期(G0)表达。为了完成这篇文章,首先,在预定的时间步长将每个孔的培养基完全去除,并用PBS洗涤每个支架两次。然后,将支架在4%多聚甲醛的PBS中室温孵育1小时,使细胞固定。固定结构在含有0.1-0.25%Triton X-100的PBS中渗透15分钟,用PBS洗涤2次。之后,用3% BSA/0.1% Triton X-100在PBS中阻断 细胞1小时,然后在PBS/1% BSA中加入浓度为5µgmL-1的Anti-Ki67抗体,在4℃孵育过夜。然后,在加 入山羊抗兔IgG (H+L)二抗之前,用PBS/1% BSA清洗3次,5分钟,孵育2小时。然后,在加入Hoechst 33,342(孵育1小时)之前,用PBS/BSA重新清洗2次,5分钟。z后,使用激光扫描共聚焦显微镜对细胞 进行成像。
计算方法
细胞自动机模型描述。该模型将时间模拟为离散的、均匀的时间步长;每个时间步长为1 h,每次模 拟11天。该模型模拟了采用生物3D打印方法制备的多孔细胞负载支架的子域,该子域由190 × 190×30晶格点的方形晶格组成,对称地由4个宽度为50 × 50 ×30晶格点的孔隙组成。水凝胶内的每个 晶格点可以被细胞占用或保持空缺,而孔隙中的网格点应该保持未被占用,因为相应的体外实验中的 细胞不会移动到孔隙中(Supplementary Material,图S2)。通过在水凝胶晶格上的随机位置放置指定 初始数量的细胞来实例化模拟。在每个时间步,细胞的行为都遵循一组描述细胞过程的随机规则,如 增殖、运动和死亡。生物打印中每个时间步内的主要算法和进程安排如图9所示。计算框架建立在先 前的理论细胞群工作之上40,41。使用概述、设计概念和细节(ODD)协议制定的详细模型描述,在补充材 料(补充材料,硅片研究)中提供。
细胞增殖过程。这个过程是模拟每个细胞单独考虑细胞之间的变化。单个细胞的特征是一个特定的, 随机的倍增时间,这归因于每个细胞完成一个细胞周期分裂所需的时间。根据我们的实验数据,每个细胞的倍增时间从正态分布中选取,其值为µ=96 h,标准差为 σ=6 h。模拟的细胞增殖周期过程包括增殖期和非增殖期(G0)之间的过渡。分裂后,亲本细胞保持在当前位置,子细胞可以放置在亲本细胞 附近的一个未占用的晶格点上。如果每个相邻的位点都已被占用,亲本细胞进入g0期,即静止或静止 期,此时细胞变得不活跃42 。一阶、二阶和三阶摩尔邻域用于放置子细胞。支架具有指定的z大承载 能力C来容纳细胞,当达到该容量的细胞总数时,增殖以指定的概率(P0)终止。承载能力取决于体外 系统的空间和营养限制。C和P0的值根据体外观察进行校准。由于支架内营养物质和氧气分布不均,一些细胞在达到支架承载能力后仍能增殖,而另一些细胞则进入G0期。这意味着细胞可以在有足够的 营养和自由邻近晶格点的地方继续增殖。表S1包含了所有芯片参数的值。
运动的过程。在运动过程中,细胞每移动一个特定的时间,称为 mc,但由于所有细胞的运动可能不同 步,因此引入一个随机数添加到mc中。mc参数的值也使用体外数据校准。在这个过程中,单个细胞可 以以随机概率的随机方式移动,或者以偏倚概率的偏倚随机方式移动,如果在它们的邻居中有一个自由的晶格点,它们就会改变它们的位置34。细胞可以以相同的概率向邻近的六个方向 (右、左、上、下、前、后)中的一个方向移动,这被称为随机运动,或者以加权概率的偏随机方式移动,其中细胞被 其他细胞和附近的毛孔吸引,这是由体外实验的经验观察所激发的。在偏随机运动中,我们计算每个 方向(direction-p)的运动概率,这取决于一个细胞在其吸引范围内该方向的邻近细胞的数量,定义 为(Lc)。此外,每个方向的概率可以根据个体与特定吸引力范围内的孔隙之间的欧几里得距离 (Lp)而 增加。在随机移动情况下,Pup = Pdown = Pleft = right = Pforward = Pbackward = 1/6 ,其中6 为方向数,Pup、Pdown、Pleft – right 、Pforward、Pbackward分别为细胞向上、向下、向左、向右 移动的概率。在随机偏置情况下,Pup = P1, Pdown = P2, right = P3, Pleft = P4, Pforward =P5, Pbackward = P6 ,其中 6 i=1Pi =1。概率Pi是通过扫描和求和相邻的细胞和孔晶格点来计算 的。在补充材料中描述了更多细节。z后,每个单元格以更大的趋势向计算出更高概率的方向移动。
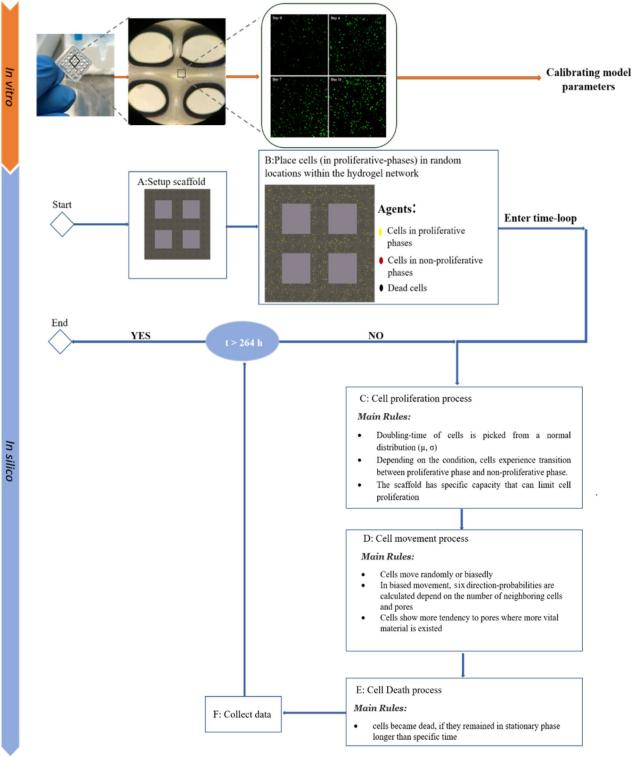
图9 。体外和硅生物打印的主要步骤。
死亡。在模拟中,我们假设3D生物打印支架的多孔结构允许所有细胞获得营养和氧气,从而防止由于 缺乏这些重要材料而导致的死亡。然而,如果细胞在固定相中停留的时间超过特定时间(定义为Cd,在一定范围内校准,概率为Pd),细胞就会失去活力。表S1包含了所有芯片参数的值。
统计分析。使用Image J软件对图像进行分析。图表和报告数据中的所有误差条均表示至少三次重复的±SD (n≥3)。结果以mean±SD格式报道。共聚焦显微镜图像的统计数据是通过平均每个结构至少 五个不同点的细胞数量获得的。
数据可用性
支持本研究结果的数据可根据合理要求从通讯作者Dorsa Mohammadrezaei处获得。
收稿日期:2022年8月13日;录用日期:2023年1月16日 出版日期:2023年1月21日
-
- Cellink 生物3D打印机 INKREDIBLE+
- 品牌:瑟林克
- 型号:INKREDIBLE+

-
- Cellink 生物3D打印机 BIO X
- 品牌:瑟林克
- 型号:BIO X
-
- Cellink BIO X6 3D生物打印机
- 品牌:瑟林克
- 型号:BIO X6


