背景
我们在进行病理学研究时,常常需要从活检组织中观察细胞的形态和结构,以及细胞内和细胞间的通讯情况。但是,大多数生物材料易碎、易腐烂,因此组织样本的正确制备和保存至关重要。
福 尔马林固定、石蜡包埋(Formalin Fixed and Paraffin Embedded, FFPE)是世界各地病理实验室保存临床组织样本的标准方法,最 早可以追溯到19世纪后期。
这种方法首先通过将组织样本固定在甲醛(也称为福 尔马林)中,以保存组织内的蛋白质和重要结构。接下来,将其嵌入石蜡块中,使得切割所需尺寸的切片更容易安装在显微镜载玻片上进行检查。FFPE样本可以在常温下储存数十年不会降解或腐烂,常被用于活检标本的保存和制备,有助于检查、实验研究和诊断/药物开发。
FFPE样本可以实现什么研究目的
FFPE样本是组织学和免疫组织化学的研究材料。但是,现如今单纯研究这一层面的变化,已经很难满足系统生物学和现代医学越来越高的研究期望。那么FFPE样本中到底包含哪些可用的信息,还能够给我们的科学与临床研究带来什么价值?首先我们需要了解FFPE的处理会对样本产生哪些相关影响,这样我们就可以充分认识哪些生物信息是可以提取的:
(1) 通过福 尔马林处理的样本,DNA/RNA会与蛋白产生交联化,整体的结构会固定,因此有助于病理观察;
(2) 同时借助于石蜡包埋避免了物质与空气的接触,内部的生物信息也能长期保存;
(3) 对于后期的分析而言,解交联后的DNA/RNA会产生明显的片段化,蛋白活性也会大大减弱或者失去。
综合上述的情况,对于传统的多组学研究,包括基因组/转录组(基因组分析、表达谱分析),蛋白组(蛋白表达谱、蛋白鉴定分析),代谢组(全靶代谢物分析),以及脂质组学的研究,FFPE样本均可实现。另外作为常见的临床研究样本,借助于病理制片技术,可以对组织样本进行深入观察与精 准收集,从而实现空间信息与多组学的整合。
FFPE样本实现的组学研究
随着分子生物学相关技术的不断发展,FFPE样本在应用中的各种难题得以解决,因此FFPE样本被广泛应用于各组学研究中。
1. 空间切割技术
在组织器官中,细胞内、细胞间的信号和体液刺激作用于特定的细胞。在病理状态下,这些特定的细胞发生了复杂的分子变化,我们想要挖掘FFPE样本的组学信息就需要研究这些目标细胞。
然而,目标细胞常常仅占细胞总体积的很小一部分,而且被其他细胞所环绕,因此如何准确地分离出目标细胞至关重要。
1996年,美国国立卫生院(NIH)国家肿瘤研究所开发出激光捕获显微切割技术(Laser Capture Microdissection,LCM),成功解决了组织中细胞异质性的问题,实现更加精 准的特异性分析。
激光显微切割是一种在显微镜观察下,不破坏组织结构,从冰冻或FFPE切片中分离出特定的单细胞或某个区域组织的技术。这种方法具有分离速度快、精 准、捕获的样本形态学特征完整、无污染等优点,满足下游组学研究的需求。
丹纳赫生命科学旗下徕卡的激光显微切割系统LMD6/7,针对FFPE样本的富集和捕获单细胞测序建库有显著的优势。

徕卡激光显微切割系统LMD6/7
2. 基因组学/转录组学
我们都知道,生物体的遗传信息记录在其基因组的DNA上,然后通过转录表达出来。基因组学就是研究基因组的结构、功能、进化、定位和编辑,以及它们对生物体的影响。而转录组学则是从RNA水平研究基因的表达,在整体水平上研究基因转录的情况和调控规律:《Cell Reports》2021年报道了使用多组学方法来揭示胰 腺神经内分泌肿瘤 (PNEN)异质性背后的分子因素。作者对胰 腺神经内分泌肿瘤FFPE临床样本进行转录组学分析,确定了具有不同分子特征的四个亚组,利用全外显子组测序揭示了亚组富集的突变差异。
上面我们提到FFPE样本制备时固定这一步,容易出现核酸片段化、核酸与蛋白交联等现象,极大地影响了样品质量、核酸提取以及下游研究(包括 RNA 测序)的可行性,这也是FFPE样本在基因组学/转录组学应用中的一项巨大挑战。
为了解决这一难题,丹纳赫生命科学旗下贝克曼库尔特提供FormaPure XL FFPE样本核酸提取试剂盒,很大程度上保证了从FFPE样本中获得产物的完整性、产量和纯度。并且可搭配同品牌的Biomek i7自动化工作站,获得FFPE样本核酸提取后,目标区域捕获测序文库制备可由Biomek液体处理工作站自动化完成,减少了手工操作时间,大大提高了工作效率,提高了结果的重复性和均一性。Biomek i7搭配AMPure XP产物纯化试剂、SPRIselect片段筛选试剂进行高通量的NGS反应体系纯化,可实现从核酸提取到捕获后PCR产物纯化的整个流程。

贝克曼库尔特Biomek i7实验室自动化工作站
此外,丹纳赫生命科学旗下的IDT可提供xGen cfDNA & FFPE DNA文库制备v2 MC试剂盒和xGen broad range RNA 建库试剂盒,这两款试剂盒进一步优化了对降解样本和低频变异检测的流程,尤其适合基于FFPE样本的基因组/转录组表达谱分析。
我们还可以使用IDT的 xGen Exom hyb panel 全外显子建库试剂盒全面捕获FFPE样本的外显子DNA文库片段。也可以根据客户检测需求,定制xGen杂交捕获探针,通过IDT独有的“目标感知”(capture-aware) 算法进行设计,特异地捕获用户感兴趣的目标基因与位点,实现更高性价比的NGS检测。
IDT xGen Exom hyb panel 全外显子建库试剂盒
3. 蛋白组学/代谢组学/脂质组学
多色荧光免疫组化(mIHC)技术
显色免疫组织化学 (IHC) 通常用于确定 FFPE 组织中的原位生物标志物表达 (例如DAB染色),但存在许多固有的局限性,包括每次只分析一种靶标、非线性染色强度、以及由于主观半定量而导致的结果不稳定性。
上述这些局限性促使研究人员开发一种能够从生物组织样本中进行定量、高维、原位数据采集的分析技术,多色荧光免疫组化(mIHC)应运而生,成为简单且全面的空间蛋白组验证工具。
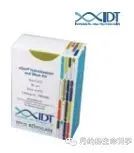
多色荧光免疫组化(mIHC)集合了免疫荧光、免疫组化和原位杂交的特点,以酪胺信号放大(TSA)为技术基础,在同一张切片上显色多个标靶,通过不同颜色来共定位蛋白质的表达。这种方法不受抗体种属的限制,具有空间定位以及定性定量分析的双重优势。尤其是在组织样本稀缺时,对每个样本收集尽可能多的数据是非常必要的:以加利福尼亚大学的研究团体的项目为例,该团体对747名结肠癌受试者的临床样本进行研究,利用多色荧光免疫组化技术在单个 FFPE 组织切片中检测到 61 种不同的蛋白质表位,揭示了结肠癌广泛的肿瘤异质性。
丹纳赫生命科学旗下的徕卡显微系统提供Cell DIVE染色成像方案,快速多标的荧光免疫组化助力FFPE样本蛋白组学的验证工作,精确、快速和灵敏度高。循环染色成像可实现一张切片超过60种蛋白的成像和分析。

徕卡Cell DIVE染色成像
液质联用(LC-MS)技术
虽然新技术的出现促进了FFPE组织中蛋白质的免疫组织学检测,但仍然有大量的蛋白质信息无法在一次实验中被检测到。此外,不同于DNA和RNA,代谢产物无法通过扩增富集,而且复杂基质中化合物种类多样,同分异构体的功能差异非常大,使得FFPE样本的代谢组学研究难以进行。
液质联用(LC-MS),全称为液相色谱-质谱联用技术,以液相色谱为分离系统,质谱为检测系统。液质联用技术体现了色谱和质谱优势的互补,将色谱对复杂样品的高分离能力,与质谱具有高选择性、高灵敏度以及能够提供相对分子质量与结构信息的优点结合起来。
科研人员使用LC-MS技术对FFPE样本进行分析,在蛋白质组学/代谢组学/脂质组学中取得了不错的进展:
蛋白质组学:Rui Sun et. al 2020利用SWATH MS技术对来自58名前列腺癌与10名良性前列腺增生病人的FFPE样本,合计共204例样本,进行分析。一共检测到27,975种蛋白质型多肽与4037种SwissProt蛋白。并且通过比较分析,从中筛选到关键的生物标志物。
代谢组学:Gomez-Gomez A et.al 2021开发了一种LC-MS/MS方法来定量15名结肠癌患者的FFPE样本中的酸性代谢物,量化了40种代谢物。
脂质组学:Sylvia K. Neef et. al 2020 优化了FFPE样本的预处理手段,通过LC-MS技术在肾FFPE样本水平中实现脂质组图谱的分析。
丹纳赫生命科学旗下SCIEX的液相色谱-质谱联用系统为FFPE组织的蛋白质组学/代谢组学/脂质组学提供从全景发现、准确鉴定到精 准定量的完整解决方案。
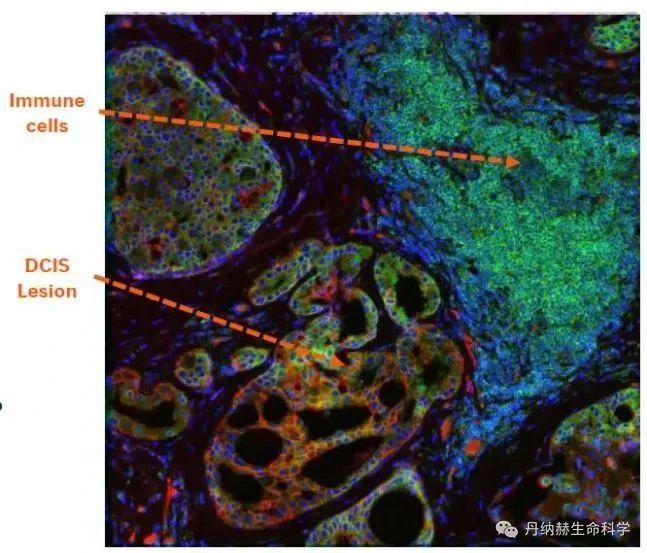
全景发现和准确鉴定:采用SCIEX ZenoTOF™串联高分辨质谱,结合Zeno™ SWATH DIA高灵敏度全景质谱采集技术,可以无偏地对蛋白质、代谢物和脂质进行质谱信号采集及差异分析。同时,利用碰撞诱导解离(CID)及电子活化解离(EAD)技术,对差异大分子及小分子进行准确鉴定及结构表征。
SCIEX ZenoTOF™串联高分辨质谱
高覆盖靶向及精 准定量分析:以SCIEX QTRAP系统为主,对特定目标组分进行分析,以验证和确认为目的,进行高通量的精 准定量分析。基于QTRAP的高灵敏度、高扫描速度和快速正负切换功能,使用Scheduled MRM方法可以在一次进样就完成1900多个脂质分子的定性和定量分析。
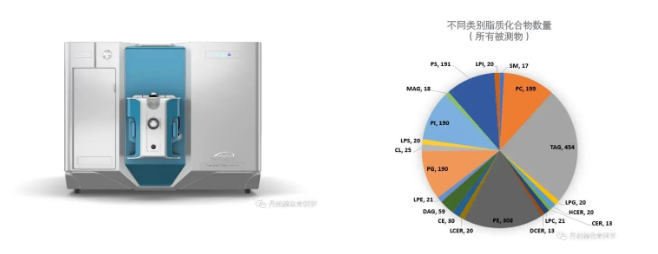
SCIEX Triple QuadTM 7500 系统
参考资料:
Guo Q, Lakatos E, Bakir IA, Curtius K, Graham TA, Mustonen V. The mutational signatures of formalin fixation on the human genome. Nat Commun. 2022 Sep 6;13(1):4487.
O'Rourke MB, Padula MP. Analysis of formalin-fixed, paraffin-embedded (FFPE) tissue via proteomic techniques and misconceptions of antigen retrieval. Biotechniques. 2016 May 1;60(5):229-38.
Hedegaard J, Thorsen K, Lund MK, et al. Next-generation sequencing of RNA and DNA isolated from paired fresh-frozen and formalin-fixed paraffin-embedded samples of human cancer and normal tissue. PLoS One. 2014 May 30;9(5):e98187.
National Human Genome Research Institute: A Brief Guide to Genomics
Yang KC, Kalloger SE, Aird JJ, Lee MKC, Rushton C, Mungall KL, Mungall AJ, Gao D, Chow C, Xu J, Karasinska JM, Colborne S, Jones SJM, Schrader J, Morin RD, Loree JM, Marra MA, Renouf DJ, Morin GB, Schaeffer DF, Gorski SM. Proteotranscriptomic classification and characterization of pancreatic neuroendocrine neoplasms. Cell Rep. 2021 Oct 12;37(2):109817. doi: 10.1016/j.celrep.2021.109817.
Gerdes MJ, Sevinsky CJ, Sood A, Adak S, Bello MO, Bordwell A, Can A, Corwin A, Dinn S, Filkins RJ, Hollman D, Kamath V, Kaanumalle S, Kenny K, Larsen M, Lazare M, Li Q, Lowes C, McCulloch CC, McDonough E, Montalto MC, Pang Z, Rittscher J, Santamaria-Pang A, Sarachan BD, Seel ML, Seppo A, Shaikh K, Sui Y, Zhang J, Ginty F. Highly multiplexed single-cell analysis of formalin-fixed, paraffin-embedded cancer tissue. Proc Natl Acad Sci U S A. 2013 Jul 16;110(29):11982-7.
Sun R , Hunter C , Chen C , et al. Accelerated Protein Biomarker Discovery from FFPE Tissue Samples Using Single-Shot, Short Gradient Microflow SWATH MS[J]. Journal of Proteome Research, 2020, 19(7).Gomez-Gomez A, Sabbaghi M, Haro N, Albanell J, Menéndez S, González M, Gil-Gómez G, Rovira A, Pozo OJ. Targeted metabolomics in formalin-fixed paraffin-embedded tissue specimens: Liquid chromatography-tandem mass spectrometry determination of acidic metabolites in cancer research. Talanta. 2021 Feb 1;223(Pt 2):121740.
Neef S K , Winter S , Hofmann U , et al. Optimized Protocol for Metabolomic and Lipidomic Profiling in Formalin-Fixed Paraffin-Embedded Kidney Tissue by LC-MS[J]. Analytica Chimica Acta, 2020, 1134.
 课堂 | 如何让样品保持在生理状态
课堂 | 如何让样品保持在生理状态

 评论
评论