比较未经处理的天然小鼠脑组织以及经固定并染色的组织在使用紫外激光切片后的RNA质量

本文介绍了样品制备过程和紫外激光显微切片(UV LMD)对小鼠脑组织冷冻切片RNA质量的影响。为在提取RNA时获取良好的结果,从高品质组织开始并在处理前后检查RNA质量至关重要。RNA完整性指数(RIN)以1到10的等级标准来显示样品质量。简言之,RIN数值越高,RNA质量越高。这项研究结果表明,可以利用紫外激光显微切片技术,可以从单个组织细胞中获取品质稳定的RNA。
评估不同制备方法
对于小脑组织切片RNA质量的影响
由于自身的不稳定性,RNA通常比DNA更难处理。RNA并不具备DNA稳定的双螺旋结构。为了在处理RNA时保证优良品质,必须从优质材料开始,并在处理前后进行特别仔细的质量控制。所谓的RNA完整性指数(RIN)是RNA质量的指标[1]。根据1到10的评价标准,RIN值为1表示RNA已完全分解,RIN值为10则表示RNA完好无损。简言之,RIN数值越高,RNA质量越高。只要可能,应始终选用具有高RIN值的材料。
固定并染色样品制备方法的影响以及后续紫外激光显微切片对于小鼠脑组织切片(事后检查)的影响描述如下。将冷冻保存且未经处理的脑组织切片的RIN值与采用标准方案处理并分离的切片进行比较。图1显示了从冷冻切片到RIN分析的样品制备工作流程。
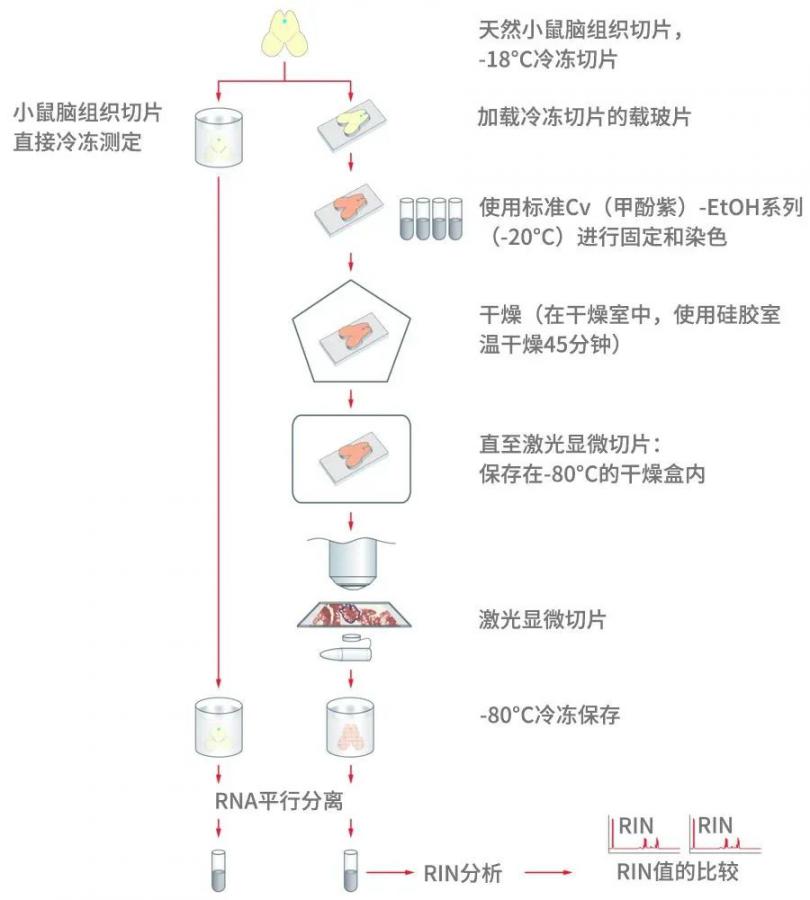
图1:小鼠脑组织从冷冻切片到激光显微切片再到RIN分析的样品处理流程。
冷冻切片,然后进行染色并固定
为了比较未经处理的组织样本与固定和染色的组织样本,使用3组已知RIN完整指数(RIN)的冷冻保存小鼠脑组织。在-80°C冷冻保存后,将它们转移到冷冻切片刀处,并在-35°C下孵育45分钟,然后将组织在-18°C下平衡30分钟,接着在-18°C下通过冷冻切片将其切成12µm的切片。小鼠脑组织切片分为两组,并按以下方式处理。一个是对照组,直接冷冻在-80°C,另一组则进行无RNase的紫外线处理PEN载玻片。这需要通过在室温下短暂解冻切片并在冷却至-20°C的75%乙醇溶液中固定2分钟来,然后,立即用几滴无菌过滤的甲酚紫(CV)染料溶液(含5%甲酚紫的纯乙醇溶液)并轻轻来回移动孵育45秒。
在染色过程中,染料溶液会短暂排除,载玻片浸入一系列浓度递增(75%、95%和100%)的乙醇溶液中,每次4秒钟,zuihou固定在100%的乙醇溶液中(试验组)。完成固定和染色程序之后,将载玻片在装有硅凝胶的干燥室中放置45分钟,然后保存在-80°C的干燥箱中,等待进行激光显微切片。
紫外激光显微切片
固定和染色之后,相关组织采用温和且无污染的紫外激光显微切片(UV LMD)进行分离[2]。这需要使用显微镜的10倍物镜对组织进行标记并分割成多个矩形,然后使用足够能量的激光束通过“Draw + Cut”(绘图切割)模式进行完全消融。切割物收集在0.5ml的无RNase盖帽中,以便对RNA内容进行提纯。这些会保存在–80°C的环境下,等待进行RNA分离。
RNA平行分离
两组样品,即保存在在 –80°C下未经处理的样品,以及经固定、染色和激光显微切片处理的样品,接受平行解冻并同时用相同的试剂处理。按照制造商的说明使用Qiagen的RNase Mini Kit®进行提纯。提纯的RNA使用30µl的无RNase水洗脱到新的无RNase试管中。
比较关系到RNA质量的RIN值
为了比较天然冷冻组织切片与经过CV染色、乙醇固定、激光显微切割的组织切片的RNA质量,将来自每个不同处理样品的RNA放入同一RNA纳米芯片(安捷伦生物分析仪)。RNA纳米芯片通过毛细管凝胶电泳分离RNA样品,并根据样品在芯片上的移动方式对样品质量进行分级。如前所述,RNA质量表示为RIN值,其中10表示完全完整的RNA,1表示完全分解的RNA。
此处进行的RIN分析(图2中的样品测量)表明,来自直接冷冻的天然组织的RNA样品的RIN值与根据UV-LMD标准方案处理的RNA样品的RIN值没有太大差异。
RNA沉淀
紧接着将分离的RNA样品分成两组,每组中有一半的样品进行沉淀。在RNA沉淀后,根据参考文件3中提供的说明,来自天然冷冻组织的已沉淀样品的RNA的RIN值略低于来自经过CV染色、EtOH固定、UV-LMD处理的样品的RNA的RIN值[3]。这种差异可能来自沉淀混合物的poly-I,因为其中一些短链RNA分子在芯片中进行RIN测量时会被提纯并与其他RNA一起检测,并计入分解部分。在qPCR 反应中,poly-I没有产生负面影响。因此,经过固定、染色以及紫外激光显微切片的脑组织切片有没有进行RNA沉淀,都不会影响其质量。图2显示了未经处理的天然小鼠脑组织切片和经过固定并染色的小鼠脑组织切片在RNA质量方面的比较结果

图2:比较未经处理的天然小鼠脑组织切片和经固定并染色的小鼠脑组织切片的RNA质量:本例显示了从天然冷冻组织(天然)和经CV染色、EtOH固定和UV-LMD切片处理的相同小鼠脑组织中分离出的RNA样品,在RNA沉淀前后的虚拟RNA凝胶图(左)和安捷伦RNA芯片的相关电泳图(右)。电泳图来自荧光单位 (FU) 随着时间的推移(以秒[s]为单位)的结果[4,5]。
结 论
总而言之,实验表明使用所述方法进行紫外激光显微切割[2],不会显著影响冷冻保存的脑组织的RNA质量。这种技术提供了一种合理的样品制备方法,并实现了单个细胞的非破坏性分离,从而获得质量一致的RNA。
相关产品

Leica LMD6 & LMD7激光显微切割
References:
1.A. Schroeder, O. Mueller, S. Stocker, R. Salowsky, M. Michael, M. Gassmann, S. Lightfoot, W. Menzel, M. Granzow, T. Ragg, The RIN: an RNA integrity number for assigning integrity values to RNA measurements, BMC Molecular Biology (2006) vol. 7, iss. 1, art. 3, DOI: 10.1186/1471-2199-7-3.
2.J. Gründemann, F. Schlaudraff, B. Liss, UV-Laser Microdissection and mRNA Expression Analysis of Individual Neurons from Postmortem Parkinson’s Disease Brains. In: Murray, G. (eds) Laser Capture Microdissection. Methods in Molecular Biology, vol. 755 (Humana Press, 2011), pp. 363-374, DOI: 10.1007/978-1-61779-163-5_30.
3.B. Liss, Improved quantitative real‐time RT–PCR for expression profiling of individual cells, Nucleic Acids Research (2002) vol. 30, iss. 17, e89, DOI: 10.1093/nar/gnf088.
4.Schlaudraff F, Gründemann J, Fauler M, Dragecivic E, Hardy J, and Liss B, Orchestrated increase of dopamine and PARK mRNAs but not miR-133b in dopamine neurons in Parkinson’s disease, Neurobiology of Aging (2014) vol. 35, iss. 10, pp. 2302-2315, DOI: 10.1016/j.neurobiolaging.2014.03.016.
5.Duda J, Fauler M, Gründemann J, Liss B, Cell-Specific RNA Quantification in Human SN DA Neurons from Heterogeneous Post-mortem Midbrain Samples by UV-Laser Microdissection and RT-qPCR, Methods Mol. Biol. (2018) vol. 1723, pp. 335-360, DOI: 10.1007/978-1-4939-7558-7_19.
 培安应邀参加ZG药物制剂高质量发展研讨会
培安应邀参加ZG药物制剂高质量发展研讨会
 逗邦®一次性使用无菌采样拭子,助力全球猴痘病毒高质量检测
逗邦®一次性使用无菌采样拭子,助力全球猴痘病毒高质量检测

 评论
评论