全场景显微成像分析平台MICA集3D采集和AI定量于一体。
3D组织成像广泛应用于生命科学领域。研究人员利用它来揭示组织组成和完整性的详细信息,或从实验操作中得出结论,或比较健康与不健康的样本。本文介绍了MICA如何帮助研究人员进行3D组织成像。
3D组织成像
模式生物或患者的组织切片可用于分析从组织到细胞的各种形态,进而发现健康和非健康样本以及对照样品和实验样品之间的差异。例如,是否存在特定细胞或它们的形态(即形状、体积、长度、面积)都是有意义的参数。
荧光显微镜有助于识别特定标记的细胞或细胞成分。因此,要么用转荧光标记基因生物,要么用免疫荧光染色。此外,某些基因和转录也可以通过荧光原位杂交 (Fluorescence in Situ Hybridization, FISH) 进行可视化。
3D组织成像的一个示例是,对脑部神经元进行成像,以确定它们的长度、体积或与其它细胞的连接。例如,可以对患有局部脑缺血的模式生物制作脑部切片,以了解形态差异和细胞数量。
挑战
首要的挑战之一是使用显微镜初步观察样本。需要将样本置于载物台上并不断调整三维位置以确保对样本进行正确成像。你从目镜或屏幕上看到的只是样本极小的一部分。因此,要将样本保持在正确的焦距内并找到正确位置,以便找到感兴趣的区域,是一个非常麻烦的过程。MICA的样本查找功能通过将样本聚焦并生成每个相关区域的低倍率预览图来自动化这个过程,这个功能可以用于整个成像过程的定位。
下一个挑战是设置成像参数,因此可以在看到感兴趣的信号下,避免样本遭受不必要的光漂白。这一步骤通常要同时选择激发和接受检测的技术参数,因为每一项参数都会对样本和获得的结果产生不同的影响。使用MICA,您只需轻轻点击一下“Live”,便可自动完成可视化荧光所需的所有参数设置。可随时通过点击“OneTouch”执行这一自动化设置来优化当前视图的参数。更改显微镜的特定技术参数前,实验人员通常需要了解更改参数将产生的影响,但在MICA中,设置是输出驱动型的,也就是说,可定义所需的输出,然后自动完成对应的调整。
一般而言,第一步是确定要成像的正确位置。实验人员需要使用目镜了解样本的整体概况,并记住不同的位置。数字显微镜可以生成样本的概览,这可以提供一些帮助,但实验人员仍然需要指出图像中要进一步成像的位置。MICA的Navigator工具可简化这一过程。用户可以生成低倍或高倍的预览,轻松定位感兴趣的区域,并可以使用工具直接在图像上标记出感兴趣的样本区域。这样后续高分辨率图片就可以保存下来。
高放大倍数物镜通常需要使用浸没式介质,最常见的是水和油。水为水溶液中的成像样品匹配了最佳的光学指数,而油为包埋的成像样品匹配了最佳的光学指数。水浸物镜也可用于固定式样本,但会稍微影响成像质量。MICA可同时满足两种需求。水镜还具有全自动化操作的额外优势,水的浸入可以自动建立并维持。为进一步提高光学质量,一些物镜会通过校正环来补偿样本板的厚度。校正环可手动、也可自动操作。MICA配置了自动校正环功能,可实现自动优化。
相对厚度是组织切片成像的另一大挑战。厚切片会形成较多的散射光,干扰所需信号。THUNDER可减少背景模糊,为组织成像提供了一种宝贵的计算成像方法。 MICA集THUNDER于一体,可在合理的时间范围内确定感兴趣的区域,
除了类似于THUNDER的计算清除方法,共聚焦激光扫描显微术(CLSM)等光学部分也是3D组织玻片成像的一种方法。这种方法中,可获得性和可用性方面也是挑战。
除了技术设置比较复杂,共聚焦显微镜所需的培训时间一般也更长。MICA集共聚焦和宽场成像于一体,最大程度减少了成像参数设置,缩短了所需的培训时间,同时也降低了操作显微镜的技能要求。
另外,共聚焦和宽场成像模式的图像设置有相同的外观和使用感受,因此,用户无需学习两种系统的操作方法。而且,用户可随意在宽场和共聚焦两种模式间切换而无需在两种成像系统间转移样本。
科学实验的一个关键方面是,改变尽可能少的变量,以确定对样本和结果的任何影响。除了保证样本处理相同外,另一个方面是针对激发和接收检测成像参数相同。MICA默认在不同项目中保持成像参数不变,用户仅基于自己的需求进行调整。可根据参考图像轻松恢复成像参数。
方法
三个厚度为250µm的小鼠脑部切片包含下述荧光标记物:
· 细胞核(DAPI,品红色)
· 神经元(细胞质GFP,青色)
· 星形胶质细胞(GFAP-DsRed,红色)
将切片固定于载玻片支架中(图1)并置于载物台上进行成像。
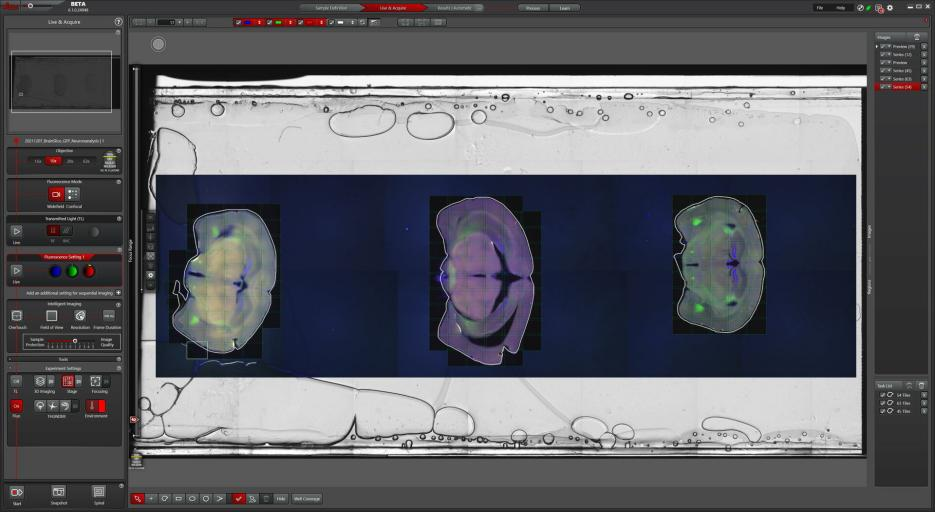
图2: MICA在实验开始时进行完整的玻片预览(宽场),便于更轻松地定位。
借助该信息的信息,可找到大图扫描拼接的感兴趣区域。可使用Magic Wand工具自动化检测感兴趣区域。
MICA可同时采集最多四个荧光团,因此相比基于滤光块的序列成像的显微系统,可有效节约用户的时间。在单次扫描拼接中,可找到感兴趣区域,并在共聚焦模式下以更高的放大倍数观察更多的细节。
二维图像需要借助三维数据以获得更详细的信息。为此,z界面中定义了三维立体模式。
在CLSM下进行立体采集后(120µm厚),可在三维观察器中可视化数据,获得脑部样本的更多空间信息。
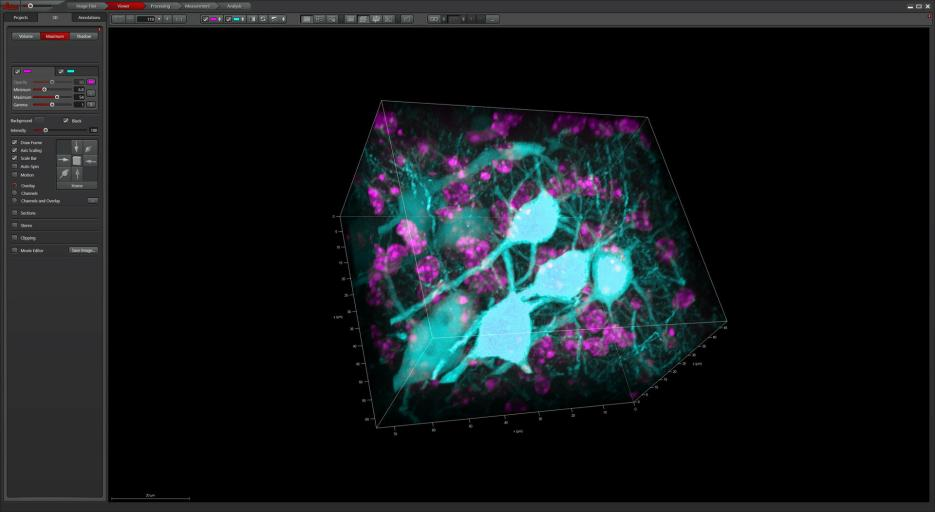 图3:三维重构CLSM。
图3:三维重构CLSM。
通过三维采集进一步研究组织切片。利用获得的三维信息,用户可以更好地了解样本的空间状况,例如了解细胞间的连接。
对于定量来说,可根据三维采集信息生成最大投影来测量样本树突棘的平均面积。pixel classifier识别棘突,分析工具则确定面积。得到的数值可绘制成图,以可视化数据和相关性。图4显示了树突棘面积的直方图。这些结果也可通过箱线图的形式显示,来比较不同的树突棘群落(图4)。
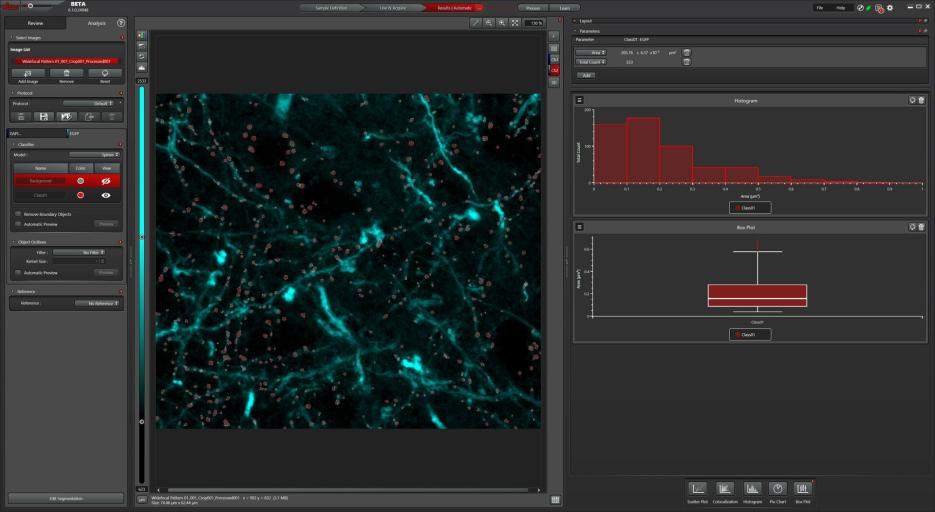
图4:分析。
MICA不仅采集图像,还可对它们进行分析。为此,可使用基于人工智能技术的pixel classifier来识别相关的图像细节。随后,识别出的对象可以被量化并显示在图形中。在本示例中,树突棘的平均面积在最大投影上测量。
结论
MICA是用于三维组织成像的有效工具:使用pixel classifier功能,用户可以快速了解样本的整体质量,确定进一步的操作。随后,Navigator视图可对组织切片进行更深入的观察。Magic Wand等工具用于快速定义感兴趣的区域,加上4个通道的同时成像,可加快大图扫描拼接的速度。使用新的z界面使三维采集更加简化,pixel classifier能辅助后续分析。
简而言之,MICA集宽场成像和共聚焦成像于一个系统中。它可以帮助用户在一个系统中完成从图像预览到三维细节成像再到分析的整个工作流程。

 评论
评论